Connectomics
Har varit i Seoul för invigningen av Korea Sweden Carbon Based Nanostructure Research Center (KSCNR). En och en halv dag av föredrag med anknytning till nanostrukturer och framförallt kolnanorör. Det bästa föredraget var dock ganska off-topic; Prof. Sebastian Seung från Howard Hughes Medical Institute och MIT talade om ”Toward a new science of connectomics”. Det ligger som sagt utanför mitt område även om Seung är fysiker i grunden. Kanske mer något för Malin på Vetenskapsnytt.
Connectome är ett nytt av en stor mängd -ome-ord, något som skall designera en helhet av något, och som blivit plågsamt ofta använt sedan HUGO-projektetet (HUman Genome Organistaion) blev så framgångsrikt. Connectomics handlar om att kartlägga anslutningarna mellan hjärnans nervceller, neuron. Det är en mäktig uppgift. En mänsklig hjärna har ungefär 1011 nervceller, och varje cell har uppskattningsvis ca tiotusen anslutningar eller axon. Det gör runt 1015 axon. Problemet med att avbilda och följa dem ligger i att axonen är tunna, bara runt hundra nanometer, vilket gör dem till några av de finaste beståndsdelarna av en levande organism, och att axonen kan ringla sig upp till flera millimetrar genom hjärnan. För att kunna avbilda och sedan följa axonen genom hjärnan och indentifiera vilka celler som kommunicerar med vilka så behöver man en teknik med en upplösning där varje pixel är mindre än ca 20nm och man behöver dessutom volymavbildningsegenskaper. 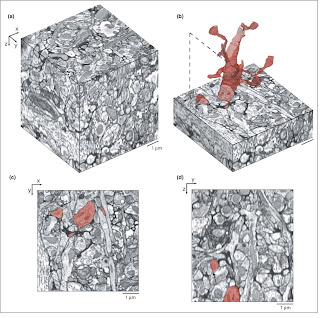 En teknik, som utvecklats av Kevin Briggman och Winfred Denk på MPI i Heidelberg innebär att man tillverkar ett fixerat block av hjärnvävnad som man färgar med ämnen som ökar kontrasen mellan olika celldelar och intercellulära delar. Detta i sig är en hel vetenskap. Sedan avbildar man ytan på blocket med svepelektronmikroskop (SEM), varefter man mha diamantkniv skär bort en 30nm tjock skiva och sedan avbildar man ytan igen osv1. Sedan låter man en dator rekonstruera blocket och identifiera de olika elementen.
En teknik, som utvecklats av Kevin Briggman och Winfred Denk på MPI i Heidelberg innebär att man tillverkar ett fixerat block av hjärnvävnad som man färgar med ämnen som ökar kontrasen mellan olika celldelar och intercellulära delar. Detta i sig är en hel vetenskap. Sedan avbildar man ytan på blocket med svepelektronmikroskop (SEM), varefter man mha diamantkniv skär bort en 30nm tjock skiva och sedan avbildar man ytan igen osv1. Sedan låter man en dator rekonstruera blocket och identifiera de olika elementen.
Det är här Seungs grupp kommer in. Datoranalysen som skall till är inte bara svår; datorer är inte särskilt bra på att identifiera mönster som ofta är inkompletta pga av varierande kontrast och andre artefakter; datamängderna är också enorma. Seung visade en video av en rekonstruktion som gjorts av ett 20x20x50µm3 stort (litet!) block. Det hade gjorts genom att man för hand gått igenom och markerat en del av volymens innehåll och låtit ett självlärande program gå igenom samma del och sedan komplettera det hela. Blocket som Seung visade innehåller ungefär 220 miljoner volymselement (voxlar) och platsar inte riktigt på en CD-skiva.
Hittills har man katalogiserat connectomet för en art, nematodmasken C. elegans. Den har 302 neuron och ca 7000 synapser, se WormAtlas. Arbetet gjordes för hand och tog runt 15 år. Nästa steg är fruktflugan Drosophila som har runt 105 neuron och runt 107 synapser. Blocket som Seung visade är ungefär en tusendel av fruktflugans hjärna. För det ultimata steget, den mänskliga hjärnan kommer man att behöva vänta på datorutvecklingen. Bara lagringsutrymmet som skulle behövas uppskattades till runt 5x1019 byte, eller några 1000 gånger mer än Googles lagringsutrymme.
Sådana här massiva datainsamlingsoperationer är egentligen ett av två vanliga sätt att göra vetenskap; antingen använder man de instrument man har eller kan utveckla till att plocka fram så mycket data som möjligt (ex HUGO), eller också så ställer man upp en hypotes som man sedan letar efter data som stöder eller falsifierar. De senare forskarna kan då god nytta av de stora databaserna.
Jag tycker det är spännade med de här stora projekten, som innebär att forskare från olika fält tvingas samarbeta, och som också är så avancerade att de tvingar fram en utveckling av ny teknik för att de skall kunna genomföras. Kanske är det här och inte i det direkta slutmålet som den stora vinsten kan ligga. Den ultimata spin-off-teknologin är ju www som kom till för att hantera de stora datamängder som man fick ut från CERN.
1 K. L. Briggman, W. Denk, Curr. Opin. Neurobiol. 16, 562 (2006). Bilden därifrån.
Andra bloggar om: Vetenskap, neurologi.

2 kommentarer:
Och sedan när man har den där stora databasen med all information om hur hjärnan är kopplad ska man också kunna gå åt andra hållet och extrahera kunskap och förståelse ur sina data. Verkligen inte lätt...
Framgår det av avbildningen om kopplingarna är inhibitoriska eller excitatoriska? Och av vilken subtyp? (Jag skulle gissa att nej, det gör det inte.) Utan den informationen kommer själva connectomet vara av betydligt mindre nytta, för att det blir svårt att säga vilken inverkan kopplingarna kommer ha. (Sedan behöver man också på sikt veta vilka ämnen som finns inne i cellen - i princip, proteomet - och i hur stora koncentrationer de finns, etc...)
Men det är ett häftigt projekt. Och jag ser fram emot att få se kartan över kopplingarna i fruktflugehjärnan, speciellt i de luktrelaterade delarna - det skulle jag kunna ha nytta av.
Jag måste erkänna att historien inte förtäljer hur mycket information man kan extrahera från SEM eller TEM-bilder. (TEM är en alternativ metod som används av konkurrerande grupper). Det kan ju vara så Seung insåg att det var en publik som var ganska dåligt insatta i detaljerna :)
Skicka en kommentar